近年来,机器学习(包括深度学习)打分函数被广泛用于预测蛋白-小分子结合自由能,并且在某些benchmark数据集上表现出优异的预测能力。但如果测试集含有与训练集相似的蛋白-小分子复合体,那么机器学习可能仅通过“学习”训练集的特定分布,就能在类似分布的测试集上取得优异表现。但这只是一种表面现象,因为这类模型往往在异分布的数据集上表现不理想,即不具备泛化能力(Generalization ability)。
基于结构的虚拟筛选的核心优势是在全新靶点上寻找新骨架小分子,因此,机器学习是否真正学习到蛋白-小分子相互作用,其打分函数是否具有泛化能力至关重要。

近日,北京生命科学研究所/清华大学生物医学交叉研究所黄牛实验室在JCIM上发表“Assessment of the Generalization Abilities of Machine-Learning Scoring Function for Structure-Based Virtual Screening”一文。黄牛团队在目前打分函数测评常用的PDBbind数据集上系统性地测试了12种用于蛋白-小分子结合自由能预测的机器学习打分函数的泛化能力,发现机器学习打分函数在新靶标上不具有预测能力。
通过可解释性分析,作者发现模型只学习到了蛋白-小分子相互作用的“表面”——小分子包埋溶剂可及表面(Buried solvent accessible surface area, Buried SASA),即Buried SASA 越大,预测的蛋白-小分子结合能越大。造成这种“表面”现象的原因是机器学习捕捉到PDBbind数据集里特定的相关性,将特定的相关性当作普适的因果性用于预测结合自由能,但这种特定的相关性却无法外推到异分布的数据集。“表面”现象再次敲响警钟,需要重视打分函数的泛化能力,采用可解释性分析来理解模型背后学习到的特征,对改进机器学习打分函数数据集和模型的构建具有指导意义。
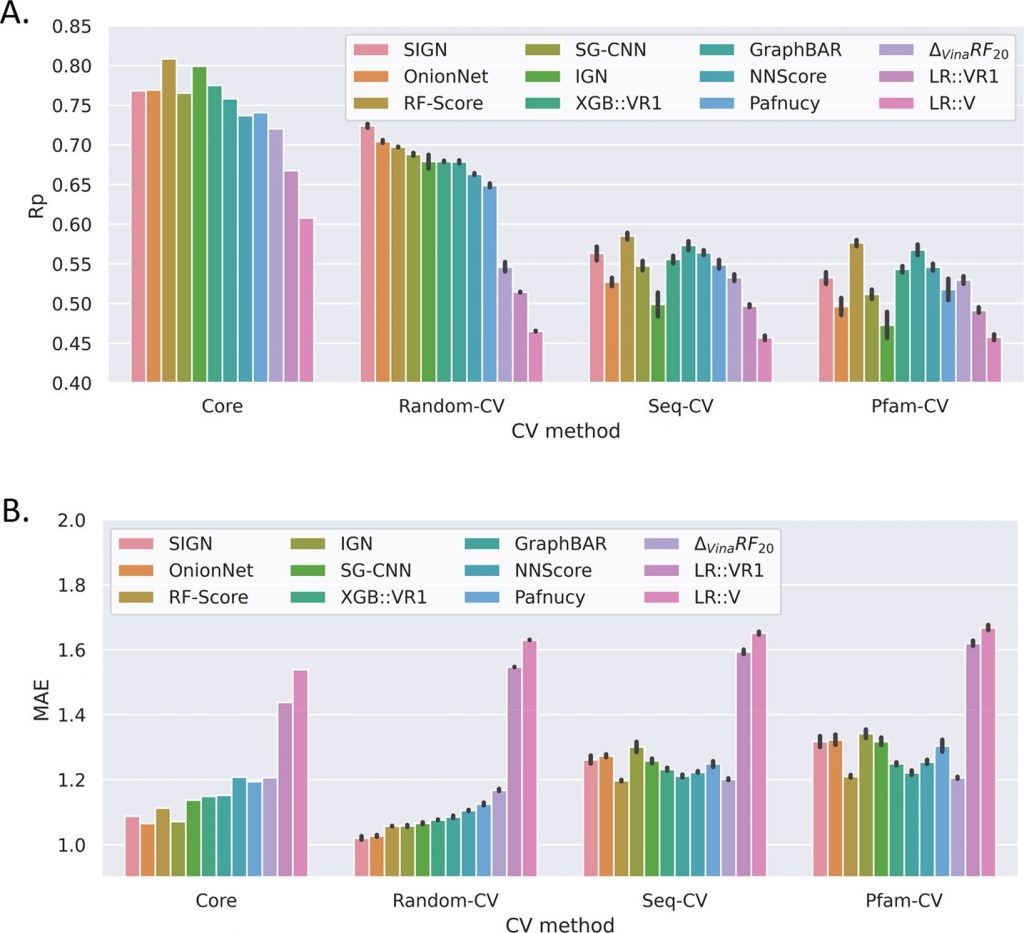
首先,作者发现传统的根据蛋白质序列相似性的方法无法合理地划分蛋白种类,导致部分与测试集相似的蛋白-小分子复合体被划分到训练集中,影响泛化能力的评估。因此,作者提出了基于蛋白-小分子结合口袋的Pfam结构域的聚类方法(Pfam cluster),对PDBbind数据集准确分类;接着测试了12种机器学习打分函数在PDBbind Core set,随机三折交叉验证(Random-CV),根据序列相似性聚类的三折交叉验证(Seq-CV),和使用Pfam cluster的三折交叉验证(Pfam-CV)上的结果。发现模型的各项指标(Rp, MAE, R2, Rs)在四次实验中依次变差,在Pfam-CV的全新的蛋白-小分子复合体上,基本不具有预测能力;并且模型越复杂,在新靶标上的泛化能力越差。相较而言,基于随机森林算法的RF-Score在新靶标上的预测能力最好。
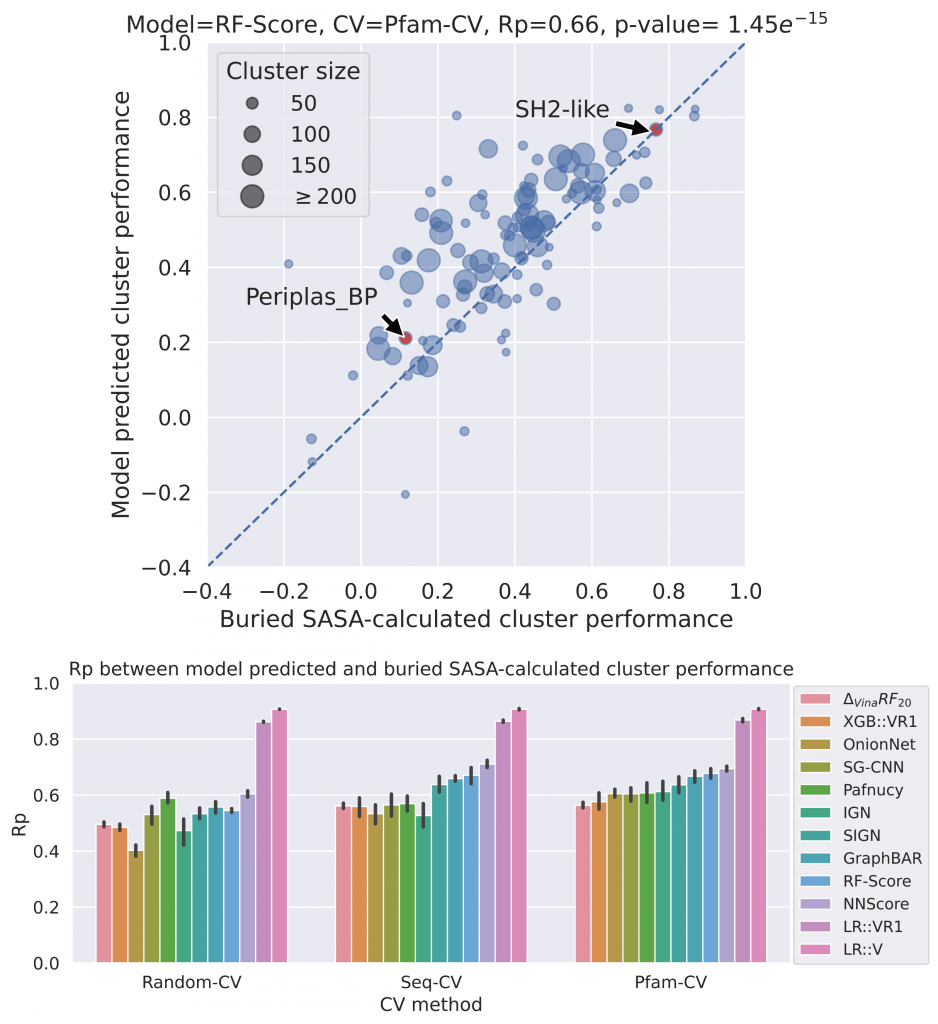
作者使用Shapley Additive exPlanations 可解释性方法分解RF-Score 42个特征的重要性,发现模型主要根据C-C(蛋白上的碳原子-小分子上的碳原子)相互作用的大小在新靶标上进行预测。因为其它模型不具有C-C相互作用这个特征,作者使用与C-C相互作用高度正相关的Buried SASA特征解释其它模型。结果发现不仅RF-Score模型,其它11个模型的预测结果都可以用Buried SASA解释——在每类蛋白(cluster)中, Buried SASA与结合自由能相关度越高(SASA相关性),模型在该类中表现越好(模型表现)。考虑所有蛋白种类,SASA相关性和模型表现呈正相关,不同模型的相关程度不同(Rp 在0.4到0.9之间),如RF-Score模型Rp为0.66。
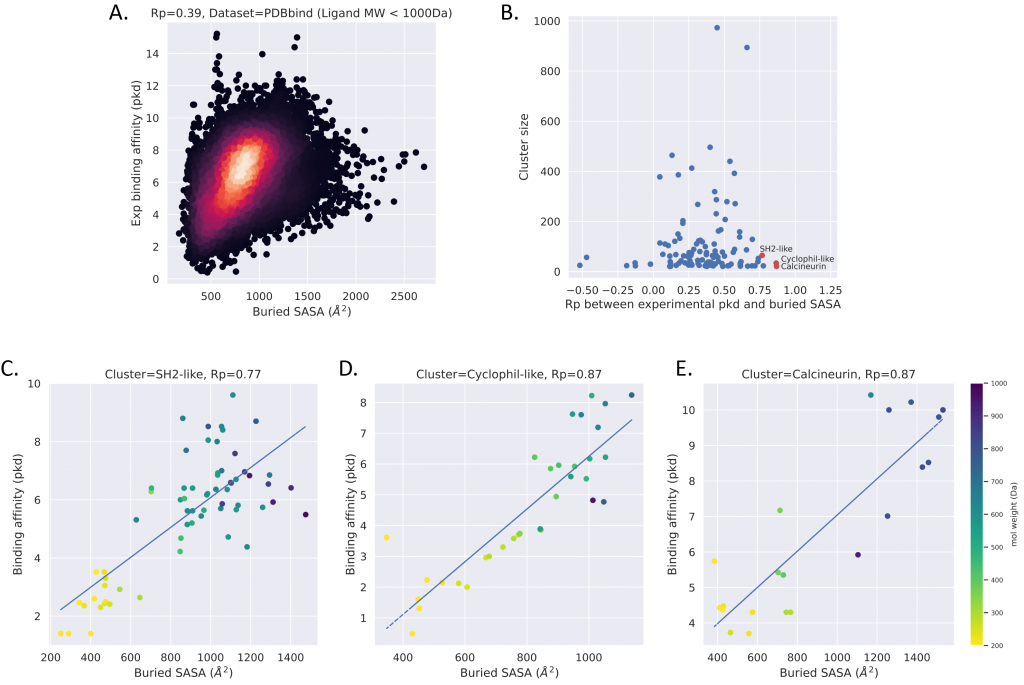
模型学习到Buried SASA这一特征的原因是模型从数据集中学习到了特定的相关性:在PDBbind数据集中,实验测得的结合能与Buried SASA整体呈正相关(Rp=0.39);尤其在某一些蛋白种类中(SH2-like, Cyclophil-like, Calcineurin),由于使用了fragment-to-lead优化策略,或含有多肽类大分子,Rp大于0.7。一般而言,机器学习算法对数据集中的相关性非常敏感,但特定的相关性并不代表普适的因果性。我们猜测因为PDBbind数据集仅包含阳性样本(活性小分子,结合足够稳定并解出复合体晶体结构),所以Buried SASA与蛋白-小分子结合能呈正相关。真实的虚拟筛选需要从海量阴性样本(无活性小分子)中挑选阳性样本(活性分子),一个仅从PDBbind数据集学会特定的SASA相关性的模型必然无法应用于真实的虚拟筛选。
综上所述,黄牛团队的这项工作透过机器学习打分函数表现优异的表面现象,看到了机器学习打分函数深藏暗处的“表面”现象。